Aminosäuresequenz ermitteln?
Hey, ich verstehe nicht ganz wie eine Aminosäuresequenz ermittelt wird. Könnte mir jemand erklären wie das funktioniert?
Zudem habe ich hier eine Aufgabe die ich machen muss, aber verstehe diese nicht ganz:
Ermitteln Sie die Aminosäuresequenzen aus den zwei angegebenen DNA-Abschnitten:
DNA-Ausschnitt aus einem codogenen Strangs des EPAS 1-Gens:
…3’-GAG TAG CTC GGA GAG CCC CGG AGC TGC AAT-5’…
DNA-Ausschnitt aus einem codogenen Strangs des EPAS 1a-Gens:
…3’-GAG TAG CTC TGA GAG CCC CGG AGC TGC AAT-5’…
Kann mit wer bei dieser Aufgabe helfen?
Danke im Voraus!
1 Antwort
Moin,
was die Aminosäuresequenzierung angeht, müsstest du schon genauer werden, denn es gibt verschiedene Methoden (Edman-Abbau; Schlack-Kumpf-Abbau; Nanoporensequenzierung; massenspektrometrische Proteinanalytik). Die alle zu erklären, ist mir jetzt zu aufwendig, nur um am Ende von dir zu hören, dass du nur Methode XY brauchtest...
Was die Translationsaufgabe angeht:
Eine DNA-Polymerase läuft einen codogenen DNA-Strang immer in 3'→5'-Richtung entlang und verknüpft bei der Transkription die RNA-Nukleotide in 5'→3'-Richtung. Dabei werden die jeweiligen Basen der DNA komplementär gepaart, wobei zu beachten ist, dass in einer mRNA anstelle der Base Thymin (T) die Base Uracil (U) eingebaut wird. Also: A mit U, C mit G, G mit C und T mit A...
Das bedeutet in deinem Fall (für das EPAS-1-Gen):
DNA-Ausschnitt:
...3’-GAG TAG CTC GGA GAG CCC CGG AGC TGC AAT-5’...
mRNA-Ausschnitt:
...5'-CUC AUC GAG CCU CUC GGG GCC UCG ACG UUA-3'...
Und diesen mRNA-Ausschnitt übersetzt du jetzt mit Hilfe einer Code-Sonne in die entsprechende Aminosäuresequenz:
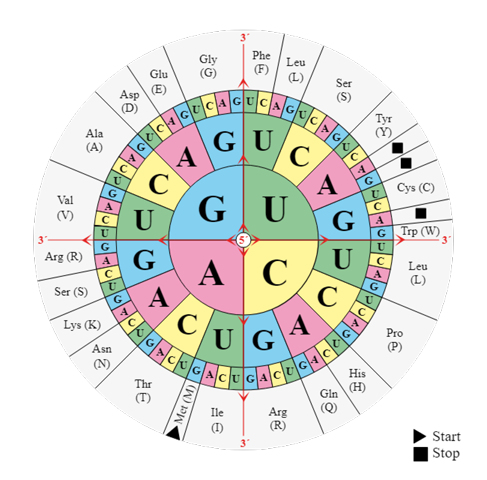
...Lys Ile Glu Pro Leu Gly Ala Ser Thr (Stopp)
So! Und nun du! Für das EPAS-1a-Gen machst du das zur Übung nun selbst.
Alles klar?
LG von der Waterkant
Vielen Dank für die ausführliche Antwort! Dadurch hat sich meine Frage eigentlich schon von selbst geklärt !