Kann mir jemand mit der folgenden Abbildung helfen?
Hey ihr, ich lern gerade auf eine Prüfung und bin bei den Altfragen auf die folgende Abb gestoßen:
Ich verstehe sie nur hinten und vorne nicht, vl kann mir jemand von euch helfen? und mir sie verständlich erklären?
Danke :)

1 Antwort
Hi,
das ist ein Klonierungsexperiment, bei dem man ein Stück DNA in einer fremden Zelle exprimieren möchte. Dazu kann man die DNA nicht einfach in die Zelle schmeißen, sondern muss sie in einen sog. Vektor einbringen, oft ein Plasmid, also eine ringförmig geschlossene DNA, die natürlicherweise auch in der betreffenden (Bakterien)Zelle vorkommt.
Die gewünschte DNA ist hier als cDNA bereits intronfrei hergestellt worden und kann in einem solchen System daher problemlos abgelesen und ausgeführt (exprimiert) werden. Wenn man das einzufügende DNA-Stück (Insert) an der EcoRI-Schnittstelle integriert, kann es sein, dass es in zwei verschiedenen Orientierungen eingebaut wird, nämlich in Leserichtung oder rückwärts. Abgelesen werden kann nur vorwärts (in Leserichtung). Um sich nun zu vergewissern, was für ein Ergebnis man vorliegen hat, kann man die Orientierung des Inserts überprüfen, indem man das Plasmid mit der eingebrachten cDNA mit weiteren Restriktionsenzymen schneidet. Dazu gibt es nur ein paar Voraussetzungen, damit es zu aussagekräftigen Ergebnissen führt:
1.) Die Schnittstelle sollte weg vom Zentrum des Inserts liegen, am besten tendenziell von der Mitte des Inserts Richtung Ende.
2.) Eine solche Schnittstelle sollte es im Insert geben und nochmals außerhalb im Plasmid.
Das ist schon alles.
Somit scheidet HindIII schon mal aus, den es liegt in der Mitte des Inserts, warum es ausscheidet, sieht man, wenn man sich eine Restriktionskarte anfertigt, eine solche für dieses Experiment habe ich einmal unten angehangen. Dazu zeichnet man sich einen Kreis, markiert darauf ca. das Insert und markiert dann eventuelle vorhandene Schnittstellen und rechnet sich dann aus, welche Fragmente beim Schneiden mit welchem Restriktionsenzym zu erwarten sind.
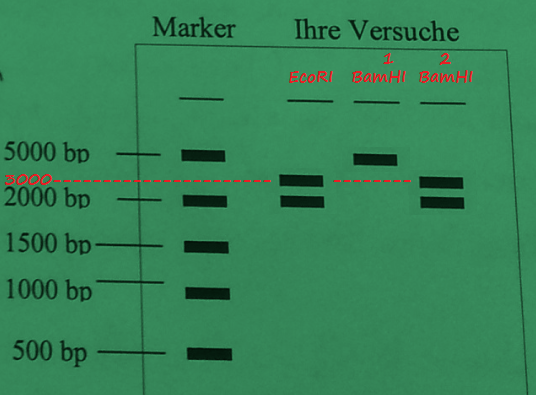
Um zu überprüfen, ob die Klonierung erfolgreich war, kann man z.B. mit dem gleichen Enzym scheiden, an dessen Schnittstelle das Insert eingefügt wurde, das wäre hier EcoRI, das führt bei erfolgreichem Einbau zu folgendem Ergebnis: 2 Fragmente der Länge 3000 bp + 2000 bp, nämlich linearisierter Vektor (Plasmid) und linearisiertes Insert, diese Fragmente laufen im Gel dann entsprechend so, dass du eine Bande bei 3000 bp beobachten kannst und eine bei 2000 bp (dazu habe ich auch eine Ergebnis-Skizze unten angehangen).
HindIII ist diagnostisch untauglich, da es das Insert in der Mitte schneidet und in der Nähe von EcoRI somit entstehen Fragmente von 4000 bp + 4000 bp, was zu einer einzigen Bande bei 4000 bp führt und somit überhaupt keine Aussage ermöglicht. Deswegen meidest du immer Restriktionsschnittstellen, die das Insert in der Mitte schneiden würden.
BamHI hingegen ist eine dankbare Schnittstelle. Sie liegt aus der Mitte des Inserts raus und es gibt sie noch einmal im Plasmid, so dass folgendes Ergebnis bei erfolgreicher Klonierung (Einbau in das Plasmid), je nach Orientierung des Inserts zu erwarten ist:
Einmal liegen die beiden BamHI-Schnittstellen nur 27 bp auseinander (+18 und -9 bp), das führt zu einem linearen DNA-Fragment von rund 5000 bp Länge, also fast dem gesamten Vektor mit Insert linearisiert. Das wäre in meiner Skizze links oder im Gel Spur BamHI 1
oder falls das Insert rückwärts orientiert eingebaut worden wäre, dann entsehen ca. 2000 bp + 3000 bp lange Fragmente, ähnlich wie mit EcoRI, weil BamHI diesmal nicht direkt neben der BamHI Schnittstelle auf dem Plasmid liegt, da das Insert anders herum eingebaut worden ist (Skizze rechts, Gel Spur BamHI 2).
Das war es eigentlich schon. D.h. auf den 3 Gelspuren kannst du die Ergebnisse von EcoRI und zwei Spuren BamHI (1+2) auftragen, nämlich einmal BamHI in die eine Richtung orientiert und die zu erwartenden Ergebnisse von BamHI, wenn das Insert andersherum integriert wurde :) Gruß, Cliff
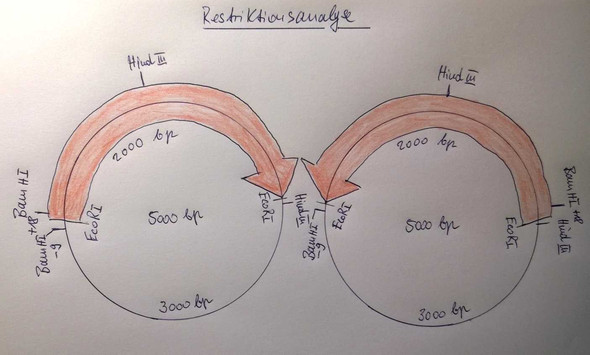
Hi, ja, da kann man nur mit den spärlichen Angaben aus der Aufgabenstellung hantieren und muss sich das zurecht basteln bzw. eine Restriktionskarte daraus basteln, wie in meiner Abb. links zu sehen. Die Angaben für die Schnittstellen müssen vorgegeben sein, sowohl für den Vektor bzw. das Plasmid, als auch für das Insert, hier die cDNA und bei dem Plasmid steht "EcoRI" sei +9bp von BamHI entfernt, wenn "BamHI" auf "0bp" liegt (unter A).
Wenn nun die eingefügte DNA an EcoRI-Schnittstelle eingefügt werden soll, wird das Plasmid mit dem Restriktionsenzym EcoRI geöffnet und -9 bp vom Insertanfang liegt BamHI, noch auf dem Plasmid.
Im Insert selbst liegt BamHi entweder +18 bp stromabwärts, wenn es so orientiert eingebaut wird, wie auf dem Arbeitsblatt dargestellt, da steht ja "EcoRI" = "0 bp" und "BamHI" = "+18 bp". In der Orientierung liegen die beiden BamHI Schnittstellen also bezogen auf den Anfang des Inserts -9 bp auf dem Plasmid und von der gleichen Stelle +18 bp auf dem Insert, macht zusammen einen Abstand von 27 bp.
Wird das Insert andersherum an EcoRI Schnittstelle eingebaut, landet die +18 bp Stelle auf der anderen Seite (vgl. meine Skizze oben, rechts). Dann liegt BamHI des Inserts 2000 bp -18 von EcoRI entfernt, das sind 1982 bp + die 9 auf dem Plasmid = 1991 bp, man kann sagen ca. ~2000 bp langes DNA Stück entsteht dann beim Schnitt mit dem Restriktionsenzym BamHI, die 9 bp kann kein Mensch im Gel unterscheiden. Aber ~2000 bp oder ca. ~5000 bp (5000 - 27 = 4973 bp) kann man schon recht gut trennen. Also die Banden tauchen deutlich unterschiedlich im Gel auf, erkennbar an den standardisierten Stücken des Markers, vgl.: https://de.wikipedia.org/wiki/DNA-Leiter Gruß
Korrektur: HindIII: 1000 bp + 4000 bp Fragmente, das kommt, wenn man Kuchen dabei mampft :) bleibt aber trotzdem unbrauchbar, da solche Fragmente bei beiden Orientierungen entstehen würden und keine Unterscheidung im Gel ermöglichen.
Wow klasse vielen vielen Dank! Hätte nicht mit so einer hilfreichen Antwort gerechnet 😀
Hey könntest du mir vl noch eine kurze Frage zu dieser Aufgabe beantworten? Wie genau erkennt man, dass bei BamHI die Schnittstelle 27bp auseinander liegt?