Biologie DNA 5' und 3'?
Hallo :) Könnte mir jemand bitte das 3' und 5' Ende beschreiben und die Bedeutung erklären?
Ich finde im Internet zwar viel, jedoch nicht direkt etwas dazu. Wäre sehr nett wenn mir jemand weiterhilft
1 Antwort

Moin, Katja...
Wie du vielleicht weißt, besteht die DNA aus aneinander gereihten Nukleotiden. Die DNA-Nukleotide ihrerseits bestehen jeweils aus
- einer der vier Basen Adenin, Thymin, Cytosin oder Guanin
- dem Zucker Desoxyribose und
- einer Phosphatgruppe (also dem Rest einer Phosphorsäure)
In organischen Molekülen (und dazu zählen hier die Purin- und Pyrimidinbasen sowie die Ribose) ist es üblich, die Kohlenstoffatome (aber auch andere Atome wie Stickstoff oder Sauerstoff, wenn sie Bestandteile von Heterozyklen sind) durchzunummerieren.
Das wird auch hier gemacht. Dabei haben die Heterozyklen der Purin- und Pyrimidinbasen die höhere Priorität. Deshalb sind hier die Kohlenstoff- und Stickstoffatome mit den Zahlen 1 bis ... nummeriert.
Um nun aber bei den Kohlenstoffatomen des Zuckers Desoxyribose, die ja auch nummeriert werden, und den Basen-Kohlenstoffen nicht durcheinander zu kommen, sind die Kohlenstoffatomzahlen des Zuckers mit einem Strich markiert. Also 1' bis 5'.

Ich habe auf die Schnelle kein Bild eines durchnummerierten DNA-Nukleotids gefunden. Oben siehst du aber das verwandte Adenosintriphosphat (ATP). Du kannst einerseits sehen, dass die Adenin-Purinbase (oben rechts) von 1 bis 9 durchnummeriert ist. Dabei hat das Stickstoffatom im Sechsring oben rechts die Ziffer 1, dann geht es im Uhrzeigersinn bis zum Kohlenstoffatom mit der Ziffer 6. Anschließend geht es weiter im Fünfring mit den Gliedern 7 bis 9.
Die Nummerierung im Zucker-Fünfring beginnt dann rechts mit der Position 1' und geht dann im Uhrzeigersinn bis 4'. Das Kohlenstoffatom des Grundgerüstes mit der Ziffer 5' ist nicht mehr Bestandteil des intramolekularen Furanoserings, sondern geht vom C4' nach oben weg (wie du siehst).
An diesem C5' sind dann im ATP-Molekül noch drei Phosphatreste (mit den Bezeichnungen alpha, beta und gamma) verestert. Die Base ist über eine sogenannte N-glycosidische Bindung mit dem anomeren C1'-Atoms der Desoxyribose verknüpft.
Ein Adenosin-Nukleotid der DNA sieht im Grunde genau so aus, nur dass es lediglich einen einzigen Phosphatrest (alpha) am C5'-Kohlenstoff gibt und anstelle der OH-Gruppe an C2'-Atom im Furanosering nur ein H gebunden ist (ohne O, deshalb ja auch Desoxyribose, also „Ribose, ohne O”...).
Wie auch immer: Die Nukleotide in einer DNA sind nun so angeordnet, dass die Basen wie halbe Sprossen einer Strickleiter zum Zentrum des DNA-Doppelstrangs gerichtet sind. Das Rückgrat bildet dagegen die Kombination aus Zucker und Phosphatrest.
Und wie du nun hoffentlich auch nachvollziehen kannst, gibt es ja in der Desoxyribose ein Kohlenstoffatom mit der Bezeichnung 3' und das schon erwähnte Kohlenstoffatom 5'.
Da am C3'-Atom eine OH-Gruppe sitzt und am C5'-Atom die Phosphatgruppe gebunden ist, die auch über eine (saure) OH-Gruppe verfügt, kann es unter Abspaltung eines Wassermoleküls (Kondensationsreaktion) zu einer weiteren Veresterung (Verknüpfung) zweier Nukleotide an diesen beiden Positionen kommen.

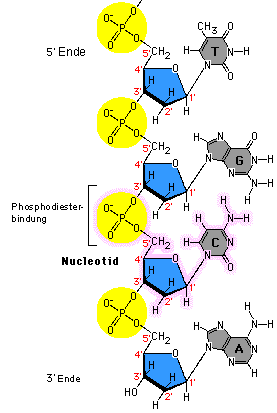
Hier siehst du einen Ausschnitt aus einem DNA-Strang mit allen Basen-Nukleotiden. Du kannst deutlich sehen, dass stets eine Phosphatgruppe zwei Nukleotide miteinander verknüpft und zwar so, dass die Phosphatgruppe eines Nukleotids über das 5'-C-Atom mit dem C3'-Atom des nachfolgenden Nukleotids verknüpft ist. Und weil diese Verknüpfung im Grunde zwischen einem Säurerest (Hydrogenphosphat; HPO4^2–) und einer alkoholischen Hydroxygruppe (–OH) erfolgte, bezeichnet man das als Esterbindung (und die Reaktion als Veresterung, denn Alkohol und Säure ergibt Ester und Wasser).
Im Bild wird das als Phosphodiesterbindung bezeichnet, weil ja quasi zwei Esterbindungen vorliegen, die zum C5' des einen und zum C3' des anderen Nukleotids.
Mit Hilfe dieser Sichtweise (und in der Darstellung oben) erkennst du nun außerdem, dass ein DNA-Einzelstrang ein 3'-Ende und ein 5'-Ende besitzt.
Dies ist die Beschreibung der Bindungsverhältnisse und wie sie zustande kommen. Die Beschreibung der DNA geht dann so weiter: Die DNA setzt sich aus zwei solcher Einzelstränge zusammen, die antiparallel zueinander sind (also einmal von oben nach unten von 3' nach 5' und einmal umgekehrt von oben nach unten von 5' nach 3' verlaufen). Die nach innen gerichteten Basen paaren sich dabei (Adenin und Thymin bzw. Guanin und Cytosin), wobei sich zwischen A und T zwei, zwischen G und C drei Wasserstoffbrückenbindungen ausbilden.

Außerdem ist die so gebildete „Strickleiter” verdrillt, das heißt sie windet sich um ihre Längsachse zu einer Doppelhelix. Man könnte noch mehr schreiben (zum Beispiel über die kleine und die große Furche oder über stabilisierende Wasserstoffbrücken der Doppelhelix), aber das soll hier mal reichen.

Zur Bedeutung der 3'- und 5'-Enden ist zu sagen, dass sie sowohl bei der Replikation (also der Verdoppelung der DNA) als auch bei der Transkription (also dem ersten Schritt der Proteinbiosynthese) eine Rolle spielen, denn die entscheidenden Hilfsenzyme bei diesen Prozessen, die Polymerasen, fahren einen DNA-Einzelstrang immer nur von einem 3'- zum 5'-Ende entlang und verknüpfen dabei die Nukleotide des zu bildenden DNA-Strangs bzw. der mRNA immer in der 5'--->3'-Richtung miteinander.
Ansonsten spielt das 5'- und das 3'-Ende noch einmal bei der mRNA eine Rolle, weil in dieser Folge die Basen-Tripletts bei der Translation (also der Übersetzung der mRNA in ein Genprodukt) zum Tragen kommen.
Wenn dir Teile dieser Ausführung zur Bedeutung der Enden jetzt noch unverständlich sind, gräm dich nicht. Das werdet ihr dann bald noch besprechen, denke ich...
So, ich glaube, das reicht, oder?
LG von der Waterkant