kann jemand mir bitte bei der aufgabe 2 helfen?

1 Antwort

Ein Kladogramm ist eine graphische Darstellung der Verwandtschaftsverhältnisse verschiedener Verwandtschaftsgruppen (Taxa, Singular: Taxon), z. B. verschiedener Arten oder Gattungen, zueinander. Es ist die einfachste Form der Darstellung von Stammbäumen. Aus dem Muster der Verzweigung des Baumes geht hervor, was mit wem am engsten verwandt ist. Im Gegensatz zu anderen Darstellungsweisen von Stammbäumen (z. B. phylogenetischen Stammbäumen) enthält ein Kladogramm keine weiteren Informationen. In phylogenetischen Bäumen sind über die Astlängen noch zusätzliche Informationen enthalten (z. B. genetische Distanz, Zeitpunkt der Aufspaltung usw.).
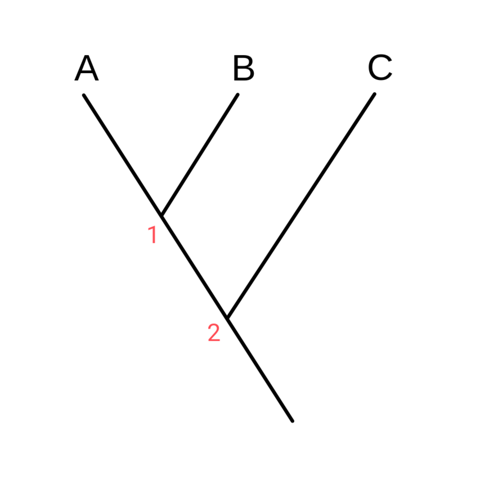
Gelesen wird ein Stammbaum immer von der Wurzel zu den Spitzen. Jeder einzelne Ast repräsentiert ein eigenes Taxon (z. B. eine Art). Die Knoten, von dem aus die Taxa sich abzweigen, symbolisieren jeweils den gemeinsamen Vorfahren. Taxa, die von einem gemeinsamen Vorfahren abzweigen, sind unmittelbar miteinander verwandt und werden als Schwester-Taxa oder Schwestergruppen bezeichnet.
Ein Beispiel soll uns dieser einfache Stammbaum von drei verschiedenen Arten (A, B und C) geben:

A und B treffen sich an einem gemeinsamen Knoten (mit 1 bezeichnet), also ist A die Schwestergruppe von B und umgekehrt.
Die Art C zweigt bereits vorher von A und B ab (Knoten 2), also ist C die Schwestergruppe eines Taxons aus A und B. Man kann das auch in Klammern im sog. Newick-Format ausdrücken (was in den Klammern steht, ist jeweils in einem Schwesterngruppenverhältnis zueinander: (C,(A,B)), C ist also die Schwestergruppe von (A,B) und umgekehrt.
In den folgenden Darstellungen habe ich das noch einmal graphisch hervorgehoben: "rot" ist jeweils die Schwestergruppe von "gelb":

sowie

Stammbäume erstellt man, indem man Merkmale der verschiedenen Taxa miteinander vergleicht. Das können morphologische Merkmale (z. B. Anzahl der Schwungfedern, Stellung der Zehen usw.) sein, aber auch Verhaltensmerkmale oder genetische Merkmale (DNA-Sequenzen oder Aminosäurensequenzen).
Es gibt verschiedene Methoden, mit denen man Stammbäume erstellen kann. In unserem Fall wählen wir ein distanzbasiertes Verfahren und als Merkmale schauen wir uns die Aminosäuresequenzen des Proteins NADH-Dehydrogenase an.
Dabei gehen wir von folgender Überlegung aus: je entfernter zwei Taxa miteinander verwandt sind, umso mehr Zeit ist seit ihrer Trennung von ihrem gemeinsamen Vorfahren vergangen. Im Umkehrschluss heißt das, dass umso weniger Zeit seit der Trennung vergangen ist, je näher zwei Taxa verwandt sind. Mit der Zeit werden sich z. B. in einer DNA-Sequenz zunehmend (aufgrund unabhängig auftretender Mutationen) Sequenzunterschiede anhäufen. Logischerweise können sich umso mehr dieser Unterschiede anhäufen, je mehr Zeit vergangen ist. Je entfernter also die Verwandtschaft ist, umso größer muss die genetische Distanz sein. Das gilt auch für die Distanz der Aminosäuresequenzen, die ja auf den DNA-Sequenzen beruht.
In einem ersten Schritt stellen wir deshalb eine Distanzmatrix auf. Das ist eine Tabelle, die paarweise die Distanzen der Taxa miteinander vergleicht. In der Aufgabenstellung ist die Distanzmatrix schon aufgestellt, es ist die Tabelle in Abb. 2.
Diese musst du dir genau anschauen und beginnst damit, diejenigen Paare herauszusuchen, die die geringste Distanz zueinander aufweisen (d. h. die größtmögliche Übereinstimmung miteinander haben). Diese sind jeweils am engsten miteinander verwandt. Beispielsweise besteht die größte Übereinstimmung zwischen Rotbrustglanzköpfchen und Gelbbauchnektarvogel (95.38 %), diese werden also miteinander am engsten verwandt sein. Dasselbe gilt für Rotrücken-Zimtelfe und Kronennymphe (92.49 %).
Von hier aus musst du dich nun im Stammbaum "rückwärts" arbeiten, d. h. du gruppierst zu der Gruppe (Glanzköpfchen-Nektarvogel) diejenige Verwandtschaftsgruppe, die jeweils die nächstgeringere Distanz aufweist. Schau also, welche der Gruppen (Schwalbe oder Mauersegler) die geringere Distanz zu (Glanzköpfchen-Nektarvogel) aufweisen. Dasselbe machst du mit (Zimtelfe-Kronennymphe).
So gehst du immer weiter, bis du schließlich an der Wurzel angekommen bist. Du musst also einfach immer nur gruppieren, was am ähnlichsten ist. Wenn du fertig bist, kannst du ja als Bild deiner Lösung hochladen und ich schau mal, ob es richtig ist.